- Tổng quan
- Nội dung
- Tiêu chuẩn liên quan
- Lược đồ
- Tải về
Tiêu chuẩn quốc gia TCVN 13842-3:2023 Phân tích dấu ấn sinh học phân tử - Phần 3
| Số hiệu: | TCVN 13842-3:2023 | Loại văn bản: | Tiêu chuẩn Việt Nam |
| Cơ quan ban hành: | Bộ Khoa học và Công nghệ | Lĩnh vực: | Khoa học-Công nghệ , Nông nghiệp-Lâm nghiệp |
|
Ngày ban hành:
Ngày ban hành là ngày, tháng, năm văn bản được thông qua hoặc ký ban hành.
|
11/09/2023 |
Hiệu lực:
|
Đã biết
|
| Người ký: | Đang cập nhật |
Tình trạng hiệu lực:
Cho biết trạng thái hiệu lực của văn bản đang tra cứu: Chưa áp dụng, Còn hiệu lực, Hết hiệu lực, Hết hiệu lực 1 phần; Đã sửa đổi, Đính chính hay Không còn phù hợp,...
|
Đã biết
|
TÓM TẮT TIÊU CHUẨN VIỆT NAM TCVN 13842-3:2023
Nội dung tóm tắt đang được cập nhật, Quý khách vui lòng quay lại sau!
Tải tiêu chuẩn Việt Nam TCVN 13842-3:2023
TIÊU CHUẨN QUỐC GIA
TCVN 13842-3:2023
ISO/TS 20224-3:2020
PHÂN TÍCH DẤU ẤN SINH HỌC PHÂN TỬ - PHÁT HIỆN NGUYÊN LIỆU CÓ NGUỒN GỐC TỪ ĐỘNG VẬT TRONG THỰC PHẨM VÀ THỨC ĂN CHĂN NUÔI BẰNG REAL-TIME PCR - PHẦN 3: PHƯƠNG PHÁP PHÁT HIỆN ADN CỦA LỢN
Molecular biomarker analysis - Detection of animal-derived materials in foodstuffs and feedstuffs by real-time PCR - Part 3: Porcine DNA detection method
Lời nói đầu
TCVN 13842-3:2023 hoàn toàn tương đương với ISO/TS 20224-3:2020;
TCVN 13842-3:2023 do Ban kỹ thuật tiêu chuẩn quốc gia TCVN/TC/F13 Phương pháp phân tích và lấy mẫu biên soạn, Viện Tiêu chuẩn Chất lượng Việt Nam đề nghị, Tổng cục Tiêu chuẩn Đo lường Chất lượng thẩm định, Bộ Khoa học và Công nghệ công bố.
Bộ TCVN 13842 (ISO/TS 20224) Phân tích dấu ấn sinh học phân tử - Phát hiện nguyên liệu có nguồn gốc từ động vật trong thực phẩm và thức ăn chăn nuôi bằng real-time PCR gồm các tiêu chuẩn sau:
- TCVN 13842-1:2023 (ISO/TS 20224-1:2020), Phần 1: Phương pháp phát hiện ADN của bò;
- TCVN 13842-2:2023 (ISO/TS 20224-2:2020), Phần 2: Phương pháp phát hiện ADN của cừu;
- TCVN 13842-3:2023 (ISO/TS 20224-3:2020), Phần 3: Phương pháp phát hiện ADN của lợn;
- TCVN 13842-4:2023 (ISO/TS 20224-4:2020), Phần 4: Phương pháp phát hiện ADN của gà;
- TCVN 13842-5:2023 (ISO/TS 20224-5:2020), Phần 5: Phương pháp phát hiện ADN của dê.
Bộ ISO/TS 20224 Molecular biomarker analysis - Detection of animal-derived materials in foodstuffs and feedstuffs by real-time PCR còn có các tiêu chuẩn sau:
- ISO/TS 20224-6:2020, Part 6: Horse DNA detection method;
- ISO/TS 20224-7:2020, Part 7; Donkey DNA detection method;
- ISO/TS 20224-8:2022, Part 8: Turkey DNA detection method;
- ISO/TS 20224-9:2022, Part 9: Goose DNA detection method.
Lời giới thiệu
Trong thực phẩm và thức ăn chăn nuôi, việc pha trộn gian lận thịt đe dọa đến thương mại và an toàn của cộng đồng. Việc pha trộn có thể ảnh hưởng đến sự tuân thủ các quy tắc về chế độ ăn hàng ngày, sự phát triển kinh tế và ổn định xã hội. Tiêu chuẩn này đưa ra phương pháp phân tích phản ứng chuỗi polymerase thời gian thực (real-time PCR) để xác định thịt của các loài động vật từ axit nucleic có trong thành phần của thực phẩm và thức ăn chăn nuôi.
Nguyên liệu sinh học có nguồn gốc từ động vật trong thực phẩm và thức ăn chăn nuôi được phát hiện và xác định trong phòng thử nghiệm bằng các bước liên tiếp (hoặc đồng thời) sau: chuẩn bị phần mẫu thử/mẫu thử, chiết và tinh sạch axit nucleic, khuếch đại PCR và diễn giải kết quả. Tiêu chuẩn này đưa ra hướng dẫn về việc khuếch đại PCR và diễn giải kết quả, cụ thể để phát hiện ADN của lợn.
Bộ TCVN 13842 (ISO 20224) bao gồm các yêu cầu kỹ thuật mô tả các ứng dụng cụ thể. Các phương pháp phát hiện ADN của các loài khác được thiết lập trong tương lai sẽ được thêm vào như các phần độc lập.
PHÂN TÍCH DẤU ẤN SINH HỌC PHÂN TỬ - PHÁT HIỆN NGUYÊN LIỆU CÓ NGUỒN GỐC TỪ ĐỘNG VẬT TRONG THỰC PHẨM VÀ THỨC ĂN CHĂN NUÔI BẰNG REAL-TIME PCR - PHẦN 3: PHƯƠNG PHÁP PHÁT HIỆN ADN CỦA LỢN
Molecular biomarker analysis - Detection of animal-derived materials in foodstuffs and feedstuffs by real-time PCR - Part 3: Porcine DNA detection method
1 Phạm vi áp dụng
Tiêu chuẩn này quy định phương pháp phản ứng chuỗi polymerase thời gian thực (real-time PCR) để phát hiện định tính ADN đặc hiệu của lợn trong thực phẩm và thức ăn chăn nuôi. Phương pháp này yêu cầu chiết một lượng thích hợp ADN có thể khuếch đại bằng PCR từ nền mẫu liên quan và có thể được áp dụng để phát hiện nguyên liệu có nguồn gốc từ lợn nhà (Sus scrofa domesticus) và lợn rừng (Sus scrofa).
Trình tự đích là một phần đoạn gen beta actin (ACTB) của lợn rừng (Sus scrota), một đoạn gen mã hóa không đầy đủ (nghĩa là mã số truy cập trong Ngân hàng gen là DQ452569.1)[1], biểu hiện dưới dạng một bản sao trên một bộ gen đơn bội. Phép phân tích PCR được dùng cho đích này có giới hạn phát hiện tuyệt đối là năm bản sao cho mỗi phản ứng, với độ lặp lại ≥ 95 % ở nồng độ này (LOD95 %).
2 Tài liệu viện dẫn
Các tài liệu viện dẫn sau rất cần thiết cho việc áp dụng tiêu chuẩn này. Đối với các tài liệu viện dẫn ghi năm công bố thì áp dụng phiên bản được nêu. Đối với các tài liệu viện dẫn không ghi năm công bố thì áp dụng phiên bản mới nhất, bao gồm cả các sửa đổi, bổ sung (nếu có).
TCVN 7606 (ISO 21571), Thực phẩm - Phương pháp phân tích để phát hiện sinh vật biến đổi gen và sản phẩm có nguồn gốc biến đổi gen - Tách chiết axit nucleic
TCVN 7608 (ISO 24276), Thực phẩm - Phương pháp phân tích để phát hiện sinh vật biến đổi gen và sản phẩm có nguồn gốc biến đổi gen - Yêu cầu chung và định nghĩa
TCVN 11933 (ISO 16577), Phân tích dấu ấn sinh học phân tử - Thuật ngữ và định nghĩa
TCVN 13841 (ISO 20813), Phân tích dấu ấn sinh học phân tử - Phương pháp phát hiện và xác định nguyên liệu có nguồn gốc từ động vật trong thực phẩm và sản phẩm thực phẩm (dựa trên axit nucleic) - Yêu cầu chung và định nghĩa
3 Thuật ngữ và định nghĩa
Trong tiêu chuẩn này sử dụng các thuật ngữ và định nghĩa nêu trong TCVN 11933 (ISO 16577).
4 Cơ sở khoa học
ADN được chiết ra khỏi phần mẫu thử bằng phương pháp thích hợp [xem Phụ lục A.1, TCVN 7606 (ISO 21571)]. Phép phân tích ADN gồm hai phần:
- kiểm tra xác nhận chất lượng và khả năng khuếch đại của ADN chiết được sử dụng phép phân tích PCR đặc hiệu đối với sinh vật nhân thực (ví dụ: gen 18S rARN) hoặc động vật có vú và gia cầm (ví dụ: gen myostatin);
- phát hiện trình tự ADN đặc hiệu của gen actin beta đơn lẻ của lợn (ví dụ: mã số truy cập trong Ngân hàng gen là DQ452569.1) bằng real-time PCR.
CHÚ THÍCH: Số bản sao gen 18S ribosome ARN (18S rARN) của sinh vật nhân thực trong một tế bào thay đổi từ vài trăm đến vài nghìn, trong khi gen beta actin ở bộ gen của lợn, gen myostatin ở bộ gen của động vật có vú và của gia cầm là gen đơn lẻ. Số bản sao của gen beta actin ở bộ gen của lợn đã được khẳng định bằng phân tích tin sinh học ở toàn bộ gen (xem Phụ lục A) và sử dụng PCR kỹ thuật số để định lượng tuyệt đối.
5 Thuốc thử và vật liệu thử
5.1 Yêu cầu chung
Trong tiêu chuẩn này, chỉ sử dụng các hóa chất và nước loại tinh khiết phân tích đã được công nhận, thích hợp dùng cho sinh học phân tử. Trừ khi có quy định khác, các dung dịch phải được chuẩn bị bằng cách hòa tan các thuốc thử tương ứng trong nước, sau đó tiệt trùng bằng nồi hấp áp lực. Đối với tất cả các thao tác sử dụng găng tay không có bột. Nên sử dụng các đầu pipet được bảo vệ bằng sol khí (tránh ô nhiễm chéo).
5.2 Thuốc thử PCR
5.2.1 PCR master mix
PCR master mix chứa ADN polymerase bền nhiệt, đệm pH, KCl, MgCl2, uracil-ADN glycosylase (UDG) và bốn dNTP (dATP, dGTP, dUTP, dCTP] ở dạng cô đặc có thể pha loãng, sẵn có để sử dụng.
5.2.2 Oligonucleotid
Chất lượng của các oligonucleotid phải đủ để sử dụng làm mồi và đoạn dò. Xem Bảng 1.
Bảng 1 - Oligonucleotid
| Tên | Trình tự ADN của oligonucleotid | Nồng độ cuối cùng trong PCR |
| Gen beta actin của lợn là trình tự đích (mã số truy cập trong Ngân hàng gen là DQ452569.1)a | ||
| Porcine-97bp-F | 5'-CGTAGGTGCACAGTAGGTCTGAC-3' | 400 nmol/L |
| Porcine-97bp-R | 5’-GGCCAGACTGGGGACATG-3' | 400 nmol/L |
| Porcine-97bp-P | 5'-[FAM]-CCAGGTCGGGGAGTC-[NFQ-MGB]b-3' | 200 nmol/L |
| a Sản phẩm PCR = 335 - CGTAGG TGCACAGTAG GTCTGACGTG ACTCCCCGAC CTGGGGTCCC CAGCACACTT AGCCGTGTTC CTTGCACTCT CTGCATGTCC GCAGTCTGGC C - 431 - DQ452569.1. b FAM: 6-carboxyfluorescein, MGB: chất kết dính rãnh phụ (nhóm chất mang màu không phát huỳnh quang).. | ||
Porcine-97bp-F gồm các cặp base 335-357, Porcine-97bp-R gồm các cặp base 414-431 và Porcine-97bp-P gồm các cặp base 360-374 của gen beta actin của lợn rừng (Sus scrofa), một đoạn gen mã hóa không đầy đủ, có mã số truy cập trong Ngân hàng gen là DQ452569.1. Có thể sử dụng thuốc nhuộm reporter và/hoặc thuốc nhuộm quencher tương tự nếu cho các kết quả tương đương hoặc tốt hơn.
6 Thiết bị, dụng cụ
Các yêu cầu liên quan đến thiết bị, dụng cụ và vật liệu phải tuân theo TCVN 13841 (ISO 20813). Ngoài các thiết bị thông thường của phòng thử nghiệm, cần có các thiết bị sau.
6.1 Máy chu trình nhiệt real-time
Thiết bị khuếch đại ADN in vitro và thực hiện các chu trình nhiệt độ-thời gian cần thiết cho PCR. Ngoài ra, thiết bị phải có khả năng kích thích các phân tử huỳnh quang ở các bước sóng quy định và phát hiện đủ ánh sáng huỳnh quang phát xạ của chất đánh dấu huỳnh quang được dùng để thực hiện các phân tích định dạng TaqMan.
7 Cách tiến hành
7.1 Chuẩn bị phần mẫu thử/mẫu thử
Mẫu thử được sử dụng để chiết ADN phải đại diện cho mẫu phòng thử nghiệm và đồng nhất, ví dụ bằng cách nghiền hoặc đồng hóa mẫu phòng thử nghiệm thành hỗn hợp mịn. Chuẩn bị phần mẫu thử/mẫu thử cần tuân theo các yêu cầu chung và phương pháp cụ thể được nêu trong TCVN 7606 (ISO 21571) và TCVN 13841 (ISO 20813).
7.2 Chuẩn bị chất chiết ADN
Việc chiết/tinh sạch và định lượng ADN từ phần mẫu thử cần tuân theo các yêu cầu chung và phương pháp nêu trong TCVN 7606 (ISO 21571). Nên sử dụng các phương pháp chiết ADN được mô tả trong Phụ lục A, TCVN 7606 (ISO 21571).
7.3 Thiết lập PCR
7.3.1 Hỗn hợp phản ứng
Phương pháp này sử dụng tổng thể tích hỗn hợp phản ứng là 25 μL cho mỗi phản ứng PCR. Thiết lập thành phần phản ứng như trong Bảng 2. Thuốc thử phải được rã đông hoàn toàn ở nhiệt độ phòng. Mỗi loại thuốc thử phải được trộn cẩn thận và ly tâm trong thời gian ngắn ngay trước khi lấy bằng pipet. Hỗn hợp thuốc thử PCR được chuẩn bị có chứa tất cả các thành phần trừ ADN mẫu thử. Tổng lượng hỗn hợp thuốc thử PCR cần được chuẩn bị phụ thuộc vào số phản ứng cần thực hiện, bao gồm ít nhất một lượng được lấy bằng pipet để dự phòng cho phản ứng bổ sung. Số lượng mẫu thử và các kiểm chứng lặp lại theo TCVN 13841 (ISO 20813). Thiết lập các phép thử PCR như sau:
a) trộn hỗn hợp thuốc thử PCR, ly tâm trong thời gian ngắn và dùng pipet lấy 20 μL cho vào mỗi lọ phản ứng;
b) thêm 5 μL từng ADN mẫu thử (từ 20 ng/μL đến 200 ng/μL) hoặc kiểm chứng dương ADN đích hoặc kiểm chứng chiết mẫu trắng hoặc nước vào các lọ phản ứng tương ứng;
c) trộn và ly tâm trong thời gian ngắn.
Bảng 2 - Thành phần phản ứng khuếch đại
| Tổng thể tích phản ứng | 25 μL |
| ADN mẫu thử (từ 20 ng/μL đến 200 ng/μL) hoặc các kiểm chứng | 5 μL |
| 2 x PCR master mixa | 12,5 μL |
| Mồi Porcine-97bp-F, c = 10 μmol/L và Porcine-97bp-R, c = 10 μmol/L | 1,0 μL cho từng mồi |
| Đoạn dò Porcine-97bp-P, c = 10 μmol/L | 0,5 μL |
| Nước | Đến 25 μL |
| a Trong thử nghiệm cộng tác, sử dụng 2 x PCR master mix đã được tối ưu hóa có sẵn để dùng có chứa tất cả các thành phần, không bao gồm khuôn mẫu và mồi. 2 x PCR master mix chứa ADN polymerase bền nhiệt, hỗn hợp của các dNTP với dUTP và uracil-UDG để giảm thiểu ô nhiễm chéo PCR và chất chuẩn nội thụ động dựa trên thuốc nhuộm ROX. Có thể sử dụng các sản phẩm tương tự nếu cho kết quả tương đương hoặc tốt hơn. Nếu cần, có thể điều chỉnh lượng thuốc thử và chương trình nhiệt độ-thời gian. | |
7.3.2 Các kiểm chứng PCR
7.3.2.1 Yêu cầu chung
Các kiểm chứng PCR được nêu trong TCVN 7608 (ISO 24276) và TCVN 13841 (ISO 20813).
7.3.2.2 Kiểm chứng chất ức chế (phân tích gen tham chiếu)
Phải thực hiện phép phân tích PCR gen kiểm chứng tham chiếu (ví dụ: gen 18S rARN đối với sinh vật nhân thực, gen myostatin đối với động vật có vú và gia cầm) sử dụng các ADN mẫu thử để kiểm tra khả năng khuếch đại axit nucleic và đưa ra kiểm chứng để loại các kết quả âm tính giả.
7.3.3 Cài đặt máy chu trình nhiệt real-time PCR
Chuyển các lọ phản ứng đã chuẩn bị vào máy chu trình nhiệt. Các lọ phải được sắp xếp để tránh mọi thay đổi nhiệt độ có thể xảy ra ở thành lọ liên quan đến máy chu trình nhiệt real-time cụ thể. Bật chương trình nhiệt độ-thời gian.
7.4 Chương trình nhiệt-độ thời gian
Chương trình nhiệt độ-thời gian nêu trong Bảng 3 đã được sử dụng trong nghiên cứu xác nhận giá trị sử dụng. Việc sử dụng các điều kiện phản ứng và chu trình real-time PCR khác nhau cần được kiểm tra xác nhận. Thời gian biến tính ban đầu phụ thuộc vào master mix được sử dụng.
Bảng 3 - Chương trình nhiệt độ-thời gian
| Bước | Thông số | Nhiệt độ | Thời gian | Đo huỳnh quang | Số chu trình | |
| 1 | Biến tính ban đầu | 95 °C | 10 min | Không | 1 | |
| 2 | Khuếch đại | Biến tính | 95 °C | 15 s | Không | 45 |
| Gắn mồi và kéo dài | 95 °C | 60 s | Có | |||
8 Tiêu chí chấp nhận/từ chối
8.1 Yêu cầu chung
Cần sử dụng chương trình phân tích dữ liệu cụ thể đối với thiết bị real-time PCR tương ứng để xác định các sản phẩm PCR. Các kết quả khuếch đại có thể được biểu thị khác nhau, tùy thuộc vào thiết bị được sử dụng. Trong trường hợp không có sản phẩm PCR có thể phát hiện được (ví dụ: các kiểm chứng âm), kết quả phải được biểu thị là “không xác định”, “không khuếch đại” hoặc số chu kỳ phản ứng tối đa được thực hiện. Nếu xảy ra sự khuếch đại của trình tự đích ADN trong một mẫu thử (ví dụ: các kiểm chứng dương) thì có thể quan sát thấy đường cong khuếch đại hình chữ S (sigmoid-shaped).
Số chu kỳ được tính tại điểm giao nhau của đường cong khuếch đại và ngưỡng huỳnh quang [chu kỳ ngưỡng (Ct) hoặc chu kỳ định lượng (Cq)].
Nếu dữ liệu đo huỳnh quang không điển hình thì việc diễn giải tự động không cho kết quả có nghĩa, có thể cần thiết lập thủ công đường nền và ngưỡng trước khi diễn giải dữ liệu. Trong trường hợp này, áp dụng các hướng dẫn cho thiết bị cụ thể được cung cấp cùng với phần mềm diễn giải.
8.2 Xác định
Trình tự đích được xem là phát hiện được nếu:
- các mồi đặc hiệu của lợn Porcine-97bp-F, Porcine-97bp-R và đoạn dò Porcine-97bp-P tạo thành đường cong khuếch đại hình chữ S và có thể tính được giá trị Ct hoặc giá trị Cq;
- các phản ứng kiểm chứng PCR không bổ sung ADN (kiểm chứng thuốc thử PCR, kiểm chứng chiết mẫu trắng) không tạo ra sự khuếch đại;
- các kiểm chứng khuếch đại (kiểm chứng dương ADN đích, kiểm chứng chất ức chế PCR) tạo ra sự khuếch đại và giá trị Ct (hoặc giá trị Cq) dự kiến.
Các phát hiện vết được xác định bằng PCR có giá trị Ct trễ hơn giá trị Ct xác định được tại LOD95 % đích. Trong trường hợp phát hiện vết hoặc thu được các kết quả dương tính/âm tính trái ngược nhau từ các chất chiết khác nhau của cùng một mẫu thử, mẫu đó phải được thử lại. Cần chuẩn bị ít nhất hai chất chiết mới từ mẫu phòng thử nghiệm đã được đồng nhất. Tiến hành phân tích PCR ít nhất 20 lần lặp lại trên các chất chiết mới (ví dụ: phân tích PCR mười lần lặp lại đối với hai ADN chiết được, phân tích PCR bảy lần lặp lại đối với ba ADN chiết được). Trình tự đích được xem là “phát hiện được” nếu ≥ 95 % các kết quả PCR của chất chiết mới cho thấy phát hiện dương tính. Trình tự đích được xem là “không phát hiện dược” nếu < 95 % các kết quả PCR của chất chiết mới cho thấy phát hiện dương tính.
9 Tình trạng xác nhận giá trị sử dụng và tiêu chí hiệu năng
9.1 Yêu cầu chung
Xác nhận giá trị sử dụng theo một quy trình gồm hai phần:
a) xác nhận giá trị sử dụng nội bộ;
b) xác nhận giá trị sử dụng bằng thử nghiệm cộng tác.
9.2 Độ vững
Độ vững (robustness) của phương pháp đã được khẳng định trong thử nghiệm cộng tác bằng cách thay đổi các điều kiện phản ứng đối với các yếu tố sau:
a) thiết bị real-time PCR (ví dụ: ABI 7500, BioRad CFX96, ABI 7900 HT Fast, Eppendorf Realplex 41));
b) thể tích phản ứng: 19 μL hoặc 21 μL hỗn hợp thuốc thử PCR cộng với 5 μL ADN mẫu thử (nồng độ từ 20 ng/μL đến 200 ng/μL);
c) nhiệt độ gắn mồi: 59 °C và 61 °C;
d) nồng độ mồi hoặc đoạn dò: đều giảm 30 %.
Đối với mỗi yếu tố được thử nghiệm, các phản ứng PCR được phân tích ba lần, mỗi lần 20 bản sao của trình tự đích và 100 bản sao của trình tự không phải đích là các kiểm chứng âm. Hiệu năng của phương pháp vẫn đạt yêu cầu đối với cả các mẫu thử và các kiểm chứng âm trong các điều kiện thay đổi đối với từng yếu tố thay đổi.
9.3 Độ tái lập
Độ tái lập của phương pháp được kiểm tra xác nhận trong thử nghiệm cộng tác có 13 phòng thử nghiệm tham gia, do Trung tâm Kỹ thuật Kiểm dịch Động vật, Thực vật và Thực phẩm, Hải quan Thượng Hải[2] tổ chức, phù hợp với nguyên tắc IUPAC[3] và các hướng dẫn BVL[4]. Các phòng thử nghiệm tham gia nhận được 12 mẫu ADN để đánh giá tỷ lệ dương tính giả và âm tính giả. Tất cả các mẫu được dán nhãn với số mã hóa ngẫu nhiên và bao gồm sáu mẫu lặp lại. 12 mẫu ADN là:
- sáu lọ dung dịch ADN của lợn, 10 bản sao/μL;
- sáu lọ dung dịch ADN của ngựa, 20 bản sao/μL.
Sử dụng phương pháp real-time PCR nêu trong tiêu chuẩn này và dãy các dịch pha loãng plasmid ADN có chứa trình tự đích để xác định số bản sao. Nồng độ của plasmid-ADN (bản sao/μL) được đo bằng máy PCR kỹ thuật số.
Các phòng thử nghiệm tham gia nhận được PCR master mix và các oligonucleotid (các mồi và đoạn dò) từ đơn vị tổ chức thử nghiệm cộng tác để tiến hành các thử nghiệm PCR.
ADN bộ gen của lợn và ngựa được chiết lần lượt từ thịt lợn và thịt ngựa, sau đó được chỉnh bằng đệm 0,2 x TE đến nồng độ danh nghĩa tương ứng là 10 bản sao/μL đối với ADN của lợn và 20 bản sao/μL đối với ADN của ngựa.
Thử nghiệm cộng tác được thiết kế để xác định tỷ lệ dương tính giả và âm tính giả. Mỗi mẫu ADN được thử bởi các phòng thử nghiệm tham gia trong một thử nghiệm PCR duy nhất với 5 μL dung dịch ADN tương ứng, sử dụng quy trình và các điều kiện nêu trong Bảng 2 và Bảng 3. Kết quả của thử nghiệm cộng tác được liệt kê trong Bảng 4.
Bảng 4 - Kết quả phép thử cộng tác
| Số phòng thử nghiệm tham gia | 13 |
| Số phòng thử nghiệm đưa ra kết quả | 13 |
| Số mẫu thử cho mỗi phòng thử nghiệm | 12 |
| Số kết quả được chấp nhận | 156 |
| Số mẫu thử được chấp nhận có chứa nguyên liệu từ lợn | 78 |
| Số mẫu thử được chấp nhận không chứa nguyên liệu từ lợn | 78 |
| Kết quả dương tính giả | 0 |
| Kết quả dương tính giả (tính bằng %) | 0 |
| Kết quả âm tính giả | 0 |
| Kết quả âm tính giả (tính bằng %) | 0 |
9.4 Độ nhạy
Giới hạn phát hiện tuyệt đối (LOD95 %) đối với phương pháp này là năm bản sao ADN. Thử nghiệm cộng tác phương pháp phát hiện ADN của lợn được thực hiện đồng thời với các thử nghiệm cộng tác phương pháp phát hiện ADN của bò, cừu và gà. Trình tự ADN đích của bò, cừu, lợn và gà được tổng hợp và tạo dòng trong vectơ pUC57 [chiều dài 2 710 bp, mã số truy cập trong Ngân hàng gen/Cơ sở dữ liệu của châu Âu về trình tự nucleotid (EMBL) là Y14837]. Plasmid PUC57-bopc đã tạo thành này (chiều dài 3 127 bp) được giải trình tự để đảm bảo rằng chỉ một bản sao của trình tự ADN đích của bò, cừu, lợn và gà được chèn vào (xem Hình 1). Không thấy có đột biến bị xóa hoặc chèn trong các trình tự được chèn (xem Hình 2). Trình tự đích của các phương pháp PCR tương ứng được đưa ra.
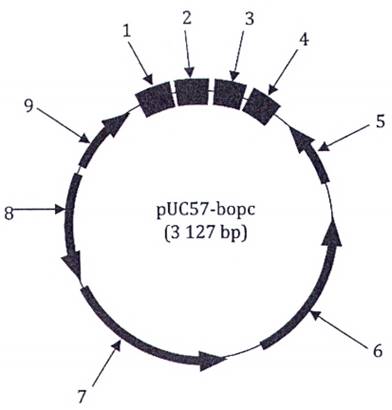
Chú dẫn:
1 nt 1~62 = amplicon bò (62 bp)
2 nt 63~150 = amplicon cừu (88 bp)
3 nt 244~340 = amplicon lợn (97 bp)
4 nt 341~417 = amplicon gà (77 bp)
5 Promoter ngược M13
6 Vị trí khởi đầu sao chép ColE1
7 Gen β-lactamase (gen kháng ampicillin)
8 Gen promoter kháng ampicillin
9 Promoter xuôi M13
Hình 1 - Sơ đồ plasmid ADN đa đích

Chú dẫn:
| Đường một gạch liền đậm | nt 1~62 = amplicon bò (62 bp) |
| Đường hai gạch liền | nt 63~150 = amplicon cừu (88 bp) |
| Đường gạch ngang | nt 244~340 = amplicon lợn (97 bp) |
| Đường gạch chấm | nt 341~417 = amplicon gà (77 bp) |
Hình 2 - Trình tự hoàn chỉnh của các nucleotide (nt) và chú giải của đoạn chèn trong plasmid pUC57
Mỗi phòng thử nghiệm tham gia vào thử nghiệm cộng tác nhận được một dung dịch chứa ADN plasmid pUC57 được chỉnh đến 1 000 bản sao/μL của trình tự đích (xem Tài liệu tham khảo [2], Hình 1 và Hình 2) trong ADN tinh trùng cá hồi nồng độ 20 ng/μL được phá vỡ tế bào bằng sóng siêu âm. Nồng độ được đo trước khi phân phối bằng thiết bị PCR kỹ thuật số (Hệ thống PCR Digital Droplet QX1002)). Dãy các dung dịch pha loãng thu được từ 13 phòng thử nghiệm nằm trong dải từ 0,02 bản sao/μL đến 4 bản sao/μL sử dụng đệm 0,2 x TE có chứa ADN tinh trùng cá hồi nồng độ 20 ng/μL được phá vỡ tế bào bằng song siêu âm. Mỗi phòng thử nghiệm tham gia đo sáu lần lặp lại cho mỗi mức nồng độ. Kết quả dương tính đạt được đối với 5 bản sao trên mỗi PCR bằng 78 trong số 78 lần thử nghiệm (xem Bảng 5).
Xác suất phát hiện (POD) mô tả xác suất mà quá trình khuếch đại PCR sẽ diễn ra tại một số bản sao đã cho của trình tự đích. Dữ liệu định tính thu được từ tất cả các phòng thử nghiệm và các mức pha loãng (xem Bảng 5) sử dụng để xác định POD = 0,95 của phương pháp phát hiện (xem Bảng 6) được nêu trong Tài liệu tham khảo [4]. Độ lệch chuẩn xác định được là 0,30 và LOD95% là 3,1 bản sao; cả hai thông số đều thấp hơn mức tối đa yêu cầu lần lượt là 1 bản sao và 20 bản sao[2][5].
Bảng 5 - Kết quả thử nghiệrn cộng tác đối với giới hạn phát hiện (LOD95%)
| Số bản sao gen beta actin của lợn trên mỗi phản ứng PCR (danh nghĩa) | Số kết quả dương tính (Ct < 45) trong 78 kết quả |
| 20 | 78 |
| 10 | 78 |
| 5 | 78 |
| 2 | 72 |
| 1 | 49 |
| 0,5 | 30 |
| 0,1 | 7 |
Bảng 6 - Kết quả thử nghiệm cộng tác đối với xác suất phát hiện (POD)
| Thông số | Gen beta actin của lợn | |
| Số phòng thử nghiệm tham gia | 13 | |
| Số lần lặp lại phân tích PCR trên mỗi mức pha loãng | 6 | |
| Đường cong POD | Xác suất phát hiện trung bình giữa các phòng thử nghiệm (LPOD) | 0,78 |
| Độ dốc b so với đường cong POD lý tưởng (b = 1) | 1,17 | |
| Độ lệch chuẩn phòng thử nghiệm, σL | 0,30 | |
| LOD95% (tính bằng các bản sao) | Trung vị của phòng thử nghiệm theo lý thuyết | 3,1 |
9.5 Độ đặc hiệu
Trình tự đại diện từ gen beta actin của lợn (mã số truy cập của trình tự trong Ngân hàng gen là DQ452569.1) được chọn làm đích PCR[1]. Mồi và đoạn dò dược thiết kế và tối ưu hóa bằng cách sử dụng phần mềm tối ưu hóa và lựa chọn đoạn dò-mồi.
Độ đặc hiệu loại trừ lý thuyết của mồi và đoạn dò gen ACTB của lợn được phân tích để xác định tính tương đồng với các loài khác sử dụng chương trình BLASTN[6]. Sử dụng trình tự 97- bp làm trình tự truy vấn là một phần của trình tự có mã số truy cập trong NCBI là DQ452569.1 (vị trí các nucleotid: 335-A31). Các kết quả nghiên cứu về sự tương đồng được nêu trong Phụ lục A. Không có sự tương đồng với các gen khác và các loài khác.
Các phép phân tích quy định trong Bảng 7 được thiết lập với ADN từ các loài khác nhau (khoảng 200 ng/PCR). Dữ liệu dự kiến lý thuyết được thiết lập bằng các trình tự truy vấn trong cơ sở dữ liệu NCBI công khai[6].
Độ đặc hiệu mục tiêu được thử nghiệm với chín giống lợn (Sus scrofa) bao gồm lợn Duroc (Mỹ), lợn Yorkshire (Anh), lợn Pietrain (Bỉ), lợn Hampshire, (Mỹ), lợn Landrace (Đan Mạch), lợn Bamei (Trung Quốc), lợn Meishan (Trung Quốc), lợn Qingping (Trung Quốc), lợn Tongcheng (Trung Quốc) và lợn rừng (Sus scrota moupinensis, Trung Quốc). Với khoảng 100 bản sao của ADN đích, tất cả các mẫu giống lợn đều được phát hiện với các tín hiệu dương tính và đường cong khuếch đại dự kiến. Độ chọn lọc mục tiêu của trình tự đích 97 base cũng được đánh giá bằng chương trình BLASTN dựa trên dữ liệu toàn bộ gen động vật của Ngân hàng gen. Các kết quả cho thấy trình tự đích 97 base là duy nhất đối với lợn được nêu trong Phụ lục A.
Bảng 7 - Độ đặc hiệu của phương pháp phát hiện gen beta actin của lợn
| Loài động vật thử nghiệm | Dự kiến về mặt lý thuyết | Khẳng định bằng thử nghiệm | |
| Động vật | Bò bison (Bison bison) | N | N |
| Lạc đà hai bướu (Camelus bactrianus) | N | N | |
| Cá chép (Cyprinus carpio) | N | N | |
| Mèo (Felis catus) | N | N | |
| Bò nhà (Bos taurus) | N | N | |
| Gà rừng lông đỏ (Gallus gallus) | N | N | |
| Chó nhà (Canis familiaris) | N | N | |
| Lừa (Equus asinus) | N | N | |
| Vịt cổ xanh (Anas platyrhynchos) | N | N | |
| Nai sừng xám (Cervus canadensis) | N | N | |
| Dê (Capra hircus) | N | N | |
| Cá vàng (Carassius auratus) | N | N | |
| Ngỗng xám (Anser anser) | N | N | |
| Ngựa (Equus caballus) | N | N | |
| Bò Zebu (Bos indicus) | N | N | |
| Chuột nhắt (Mus musculus) | N | N | |
| Khỉ vàng (Macaca mulatta) | N | N | |
| Đà điểu châu Phi (Struthio camelus) | N | N | |
| Chim trĩ đỏ (Phasianus colchicus) | N | N | |
| Lợn nhà (Sus scrofa domesticus) | Pos | Pos | |
| Bồ câu núi (Columba livia) | N | N | |
| Chim cút thường (Coturnix coturnix) | N | N | |
| Thỏ châu Âu (Oryctolagus cuniculus) | N | N | |
| Chuột cống (Rattus norvegicus) | N | N | |
| Cừu (Ovis aries) | N | N | |
| Cá hồi vân (Onchorhynchus mykiss) | N | N | |
| Gà tây (Meleagris gallopavo) | N | N | |
| Trâu (Bubalus bubalis) | N | N | |
| Lợn rừng (Sus scrofa) | Pos | Pos | |
| Bò hoang Tây Tạng (Bos mutus) | N | N | |
| Người | Người (Homo sapiens) | N | N |
| Thực vật | Cỏ linh lăng (Medicago sativa) | N | N |
| Ngô (Zea mays) | N | N | |
| Cải dầu (Brassica rapa) | N | N | |
| Lúa (Oryza sativa) | N | N | |
| Cao lương (Sorghum bicolor) | N | N | |
| Đậu tương (Glycine max) | N | N | |
| Lúa mì (Triticum aestivum) | N | N | |
| Chú dẫn Pos: dương tính; N: âm tính | |||
10 Báo cáo thử nghiệm
Báo cáo thử nghiệm phải được lập theo quy định trong TCVN 13841 (ISO 20813) và các tiêu chuẩn có thể áp dụng khác [ví dụ: TCVN 7608 (ISO 24276)].
Phụ lục A
(Tham khảo)
Kết quả BlastN 2.9.0 đối với trình tự truy vấn của bộ gen tham chiếu trong Ngân hàng gen (452 cơ sở dữ liệu)
A.1 Trình tự truy vấn
A.1.1 ID trình tự truy vấn: DQ452569.1 (bp 335-431).
A.1.2 Mô tả: gen beta actin (ACTB) của lợn rừng (Sus scrofa), một đoạn gen mã hóa không đầy đủ.
A.1.3 Loại phân tử: axit nucleic.
A.1.4 Chiều dài trình tự truy vấn: 97.
A.2 Sự phân phối 18 Blast hit trên 18 trình tự tham chiếu trong cơ sở dữ liệu của Ngân hàng gen
Xem Hình A.1.
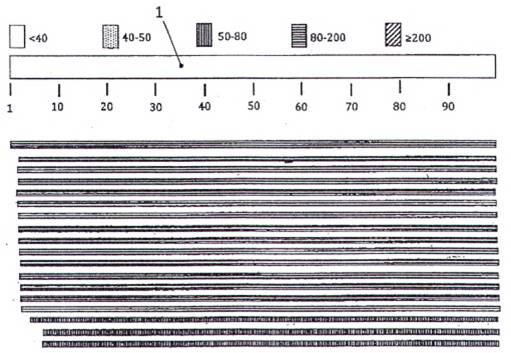
Chú dẫn:
1 Trình tư truy vấn
Hình A.1 - Chú dẫn đối với các điểm bắt cập
A.3 Mô tả
Bảng A.1 - Mô tả
| Mô tả | Điểm tối đa | Điểm tổng số | Tỷ lệ bao phủ % | Giá trị E | Độ tương đồng % | Mã số truy cập |
| Phân lập nhiễm sắc thể 3 của lợn TJ Tabasco giống Duroc loài lợn rừng (Sus scrofa), Sscrofa11.1 | 174 | 174 | 100 | 9e-40 | 98,97 | NC_010445.4 |
| Vùng gen chưa xác định của dơi quạ đen (Pteropus alecto), ASM32557v1 scaffold 40 | 145 | 145 | 98 | 7e-31 | 93,75 | NW_006431919.1 |
| Phân lập vùng gen chưa xác định của dơi Shadow, loài dơi quạ lớn (Pteropus vampyrus), Pvam_2.0 Scaffold90 | 145 | 145 | 100 | 7e-31 | 93,75 | NW_011888871.1 |
| Phân lập vùng gen chưa xác định Pink-7 của loài hươu đuôi trắng (Odocoileus virginianus texanus), Ovir.te_1.0 scaffold559 | 130 | 130 | 98 | 2e-26 | 90,82 | NW_018339053.1 |
| Phân lập vùng gen chưa xác định của lạc đà Carlotta (AHFN-0088) loài lạc đà Alpaca (Vicugna pacos), Vicugna_pacos-2.0.1 Scaffold112 | 128 | 128 | 98 | 3e-25 | 90,62 | NW_005882814.1 |
| Phân lập vùng gen chưa xác định 1219 của loài dơi ăn quả Ai Cập (Rousettus aegyptiacus), Raegyp2.0 | 126 | 126 | 98 | 3e-25 | 90,62 | NW_015494601.1 |
| Nhiễm sắc thể số 24 của chủng OAR_USU_Benz2616, giống Rambouillet, loài cừu (Ovis aries), Oar_rambouillet_v1.0 | 124 | 124 | 98 | 9e-25 | 89,80 | NC_040275.1 |
| Nhiễm sắc thể số 25 của giống dê San Clemente loài dê (Capra hircus), ASM170441v1 | 124 | 124 | 98 | 9e-25 | 89,80 | NC_030832.1 |
| Vùng gen chưa xác định của giống lạc đà Alxa loài lạc đà hai bướu (Camelus bactrianus), Ca_bactrianus_MBC_1.0 scaffold33 | 122 | 122 | 98 | 3e-24 | 89,58 | NW_011510927.1 |
| Vùng gen chưa xác định của ciống lạc đà Arabia loài lạc đà hai bướu (Camelus bactrianus), PRJNA234474_Ca_drom-edarlus_V1.0 scaffoldl638 | 122 | 122 | 98 | 3e-24 | 89,58 | NW_011510927.1 |
| Vùng gen chưa xác định của linh dương Tây Tạng (Pantholops hodgsonii), PHO1.0 Scaffold797 | 119 | 119 | 98 | 4e-23 | 88,78 | NW_005816904.1 |
| Phân lập vùng gen chưa xác định 6110/ MARCELLA của vượn cáo (Propithecus coquereli), Pcoq_1.0 Scaffold137 | 110 | 110 | 98 | 3e-20 | 87,50 | NW_012137969.1 |
| Phân lập vùng gen chưa xác định 467f Iridium của loài linh trưởng (Otolemur garnettii), OtoGar3 scaffold00092 | 110 | 110 | 98 | 4e-18 | 86,46 | NW_003852487.1 |
| Phân lập vùng gen chưa xác định ML- 2016 của dơi mũi quạ (Hipposideros armiger), ASM189008v1 | 102 | 102 | 98 | 4e-18 | 86,46 | NW_017731383.1 |
| Phân lập vùng gen chưa xác định Samal-C Ts95f của vượn mắt kính (Carlito syrichta), Tarsius_syrichta- 2.0.1 Scaffold171747 | 99 | 99 | 98 | 6e-17 | 85,42 | NW_007115843.1 |
| Phân lập vùng gen chưa xác định#75 của sóc đất 13 sọc (lctidomys tridecemlineatus), SpeTri 2.0 scaffold00297 | 75 | 75 | 96 | 1e-09 | 81,37 | NW_004936765.1 |
| Phân lập vùng gen chưa xác định SJ_83 của sóc marmot bụng vàng (Marmota fiaviventris), ASM367607v1 scaffold633 | 71,3 | 71,3 | 94 | 1e-08 | 81,00 | NW_020963434.1 |
| Vùng gen chưa xác định của sóc núi cao (Marmota marmota), marMar2.1 | 71,3 | 71,3 | 94 | 1e-08 | 81,00 | NW.015351287.1 |
A.4 Bắt cặp
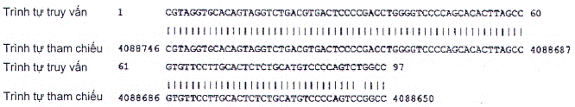
Phân lập nhiễm sắc thể số 3 của lợn TJ Tabasco giống Duroc loài lợn rừng (Sus scrota), Sscrofa11.1, ID trình tự: NC_010445.4 Chức năng: actin, cytoplasmic 1 actin, cytoplasmic 1
| Chiều dài: 132848913 | Số điểm khớp: 1 | Dải 1: từ 4088650 đến 4088746 | ||
| Điểm | Kỳ vọng | Độ tương đồng | Khoảng trống | Mạch |
| 174 bit (94) | 9e-40() | 96/97 (99 %) | 0/97 (0 %) | Dương/Âm |
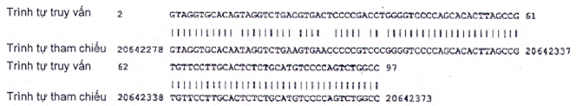
Vùng gen chưa xác định của dơi quạ đen (Pteropus alecto), ASM32557v1 scaffold 40. ID trình tự: NW_006431919,1, Chức năng: actin, cytoplasmic 1
| Chiều dài: 22055733 | Số điểm khớp: 1 | Dải 1: từ 20642278 đến 20642373 | ||
| Điểm | Kỳ vọng | Độ tương đồng | Khoảng trống | Mạch |
| 145 bit (78) | 7e-31() | 90/96 (94 %) | 0/96 (0 %) | Dương/Dương |
Phân lập vùng gen chưa xác định của dơi Shadow loài dơi quạ lớn (Pteropus vampyrus), Pvam_2.0 Scaffold90, ID trình tự: NW_011888871.1
| Chiều dài: 3778687 | Số điểm khớp: 1 | Dải 1: từ 2192495 đến 2192590 | ||
| Điểm | Kỳ vọng | Độ tương đồng | Khoảng trống | Mạch |
| 145 bit (78) | 7e-31() | 90/96 (94 %) | 0/96 (0 %) | Dương/Âm |
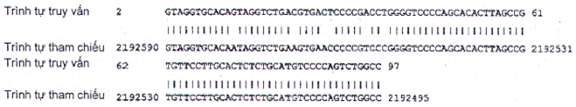
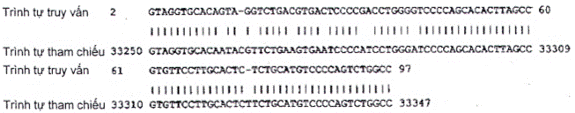
Phân lập vùng gen chưa xác định Pink-7 của loài hươu đuôi trắng (Odocoileus virginianus texanus), Ovir.te_1.0 scaffold559, ID trình tự: NW_018339053.1
| Chiều dài: 1584470 | Số điểm khớp: 1 | Dải 1: từ 33250 đến 33347 | ||
| Điểm | Kỳ vọng | Độ tương đồng | Khoảng trống | Mạch |
| 130 bit (70) | 2e-26() | 89/98 (91 %) | 2/98 (2 %) | Dương/Dương |
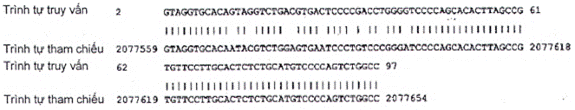
Phân lập vùng gen chưa xác định của lạc đà Carlotta (AHFN-0088) loài lạc đà Alpaca (Vicugna pacos), Vicugna_pacos-2.0.1 Scaffold112, ID trình tự: NW_005882814.1
| Chiều dài: 5706089 | Số điểm khớp: 1 | Dải 1: từ 2077559 đến 2077654 | ||
| Điểm | Kỳ vọng | Độ tương đồng | Khoảng trống | Mạch |
| 126 bit (70) | 7e-26() | 87/96 (91 %) | 0/96 (1 %) | Dương/Dương |
Thư mục tài liệu tham khảo
[1] LIN C.L., JENNEN D.G., PONSUKSILI S., THOLEN E., TESFAYE D., SCHELLANDER K., WIMMERS K., Haplotype analysis of beta-actin gene for its association with sperm quality and boar fertility. J. Anim. Breed. Genet. 2006,123(6), pp. 384-388
[2] WANG Q. CAI Y.C., HE Y.P., YANG L.T., PAN L.W. Collaborative ring trial of two real-time PCR assays for the detection of porcine- and chicken-derived material in meat products. Plos ONE. 2018, 13(10), e0206609
[3] HORWITZ W. Protocol for the design, conduct and interpretation of method performance studies. Pure and Appl. Chem. 1995, 67, pp. 331-343
[4] Guidelines for the validation of qualitative real-time PCR methods by means of a collaborative study. Federal Office of Consumer Protection and Food Safety. Bundesamt für Verbraucherschutz und Lebensmittelsicherheit (BVL)
[5] UHLIG S., FROST K., COLSON B., SIMON K., MÄDE D., REITING R., GOWIK P., GROHMANN L. Validation of qualitative PCR methods on the basis of mathematical-statistical modelling of the probability of detection. Accred Qual Assur. 2015, 20, pp. 75-83
[6] National Center for Biotechnology Information (NCBI], Available at (accessed 2019-07-28]: https://blast.ncbi.nlm.nih.gov/Blast.cgi?PROGRAM=blastn&PAGE TYPE=BlastSearch&LINK LOC=blasthome
1) Đây là các ví dụ về sản phẩm thích hợp có bán sẵn. Thông tin này đưa ra tạo thuận lợi cho người sử dụng tiêu chuẩn và không ấn định phải sử dụng sản phẩm này.
2) Đây là các ví dụ về sản phẩm thích hợp có bán sẵn. Thông tin này đưa ra tạo sự thuận lợi cho người sử dụng tiêu chuẩn và không ấn định phải sử dụng sản phẩm này. Các sản phẩm tương tự có thể được sử dụng nếu cho kết quả tương đương.
Bạn chưa Đăng nhập thành viên.
Đây là tiện ích dành cho tài khoản thành viên. Vui lòng Đăng nhập để xem chi tiết. Nếu chưa có tài khoản, vui lòng Đăng ký tại đây!
 Tiêu chuẩn Việt Nam TCVN 13842-3:2023 PDF (Bản có dấu đỏ)
Tiêu chuẩn Việt Nam TCVN 13842-3:2023 PDF (Bản có dấu đỏ) Tiêu chuẩn Việt Nam TCVN 13842-3:2023 DOC (Bản Word)
Tiêu chuẩn Việt Nam TCVN 13842-3:2023 DOC (Bản Word)